INNSBRUCK – Eine internationale Forschergruppe, darunter die Innsbrucker Mikrobiologin Rossana Segreto, hat nachgewiesen, daß es keine wissenschaftlichen Argumente gibt, daß Covid-19 nur auf natürlichem Weg entstanden sein kann und daß zentrale Argumente der Anhänger der „Natur-Theorie“ nicht haltbar sind.
Die Anhänger der Theorie, daß Covid-19 in der Natur entstanden sein könnte waren bisher ebensowenig in der Lage ihre Theorie zu beweisen, wie es die Anhänger der Labor-Theorie waren. Was jedoch auffällt ist, wie die Anhänger der Natur-Theorie mit den Vertretern des anderen Lagers umgehen.
Das Narrativ der KPC zu berechtigten Fragen: Erst „Panikmacher“, dann „Verschwörungstheoretiker“
Zu Beginn der Krise bezeichneten sie ihre Konkurrenten als „Panikmacher“; ein Narrativ, das Mitte Januar von chinesischen Behördenvertretern gegenüber der WHO ausgegeben wurde und das die Regierungen des Westens dann unkritisch nachplapperten. Nachdem dieses Narrativ von der Realität überholt worden war, bezeichneten die selben Kreise ihre wissenschaftlichen Konkurrenten einfach und völlig unwissenschaftlich als „Verschwörungstheoretiker„. Umgekehrt jedoch hätten die Anhänger der Labor-Theorie auch gute Argumente auf ihrer Seite ihre Rivalen als „Verschwörer“ zu bezeichnen, doch das geschieht nicht.
Gleich nach Amtsantritt unterzeichnete der neue Präsident der USA einen Erlass, dem gemäß es „rassistisch“ sei, Covid-19 als Wuhan-Virus zu bezeichnen. Diese Handlungen machen eines deutlich: sozialistische und kommunistische Kräfte tun alles, was in ihrer Macht steht, um die Menschen daran zu hindern, einen Bezug zwischen Cobid-19 und China herzustellen.
Diese Aufweichung durch Bidon fällt in eine Serie gleichartiger Aktionen gegenüber China.
.
Die Verstrickungen der Covid-Fachleute mit Frau Prof Shi
Einer der aggressivsten Vertreter der Natur-Theorie ist Peter Daszak. Trotz seiner evidenten Voreingenommenheit ist er Mitglied der Inspektions-Kommission der WHO, die die Vorgänge in Wuhan untersuchen soll. Daszak und auch der Chefberater zum Covid-19-Virus der USA Fauci sind mit Frau Shi eng bekannt. Man kennt sich von verschiedensten Treffen. Da stellt sich schon die Frage, warum Frau Shi nicht wenigstens ihren Kollegen Fauci und Daszak gegenüber die Virus-Sequenzen offenbart hat, um die Welt besser und schneller in die Lage zu versetzten, Gegenmaßnahmen zu treffen? Deren enger Kontakt wirft auch noch weitere Fragen auf:
Fauci hat im Dezember oder Anfang Januar nie die Nachricht erhalten, dass sich die Krankheit durch Atemkontakt verbreitet hat? Fauci war zu schüchtern, um Zhengli Shi anzurufen und nach der Genomsequenz des Virus zu fragen? Unsere Nation gab 200 Millionen Dollar für Daszaks PREDICT-Programm aus und er konnte seinem BATGIRL Zhengli Shi keine kurze SMS mit zusätzlichen Informationen senden, um die USA, FAUCI und TRUMP zu informieren. oder hat er ???
China begrüßt diese Delegation der WHO auf seine ganz eigene Weise: es löscht weitere Daten und Erkenntnisse seiner Forscher aus Wuhan.
Die zunehmenden Hinweise verdichten sich zu Beweisen
Doch diese Löschaktion könnte zu spät kommen, denn es gibt inzwischen eine große Anzahl an Veröffentlichungen durch Whistleblower, Journalisten und Wissenschaftler, die der Frage nachgehen, ob Covid-19 im Labor entstanden ist:
Am 17. November ist zu dieser großen Anzahl eine weitere wissenschaftliche Arbeit veröffentlicht worden. Diese Arbeit eines internationalen Forscherteams, an dem Rossana Segreto von der Uni Innsbruck und Yuri Deigin unter dem Titel
„The genetic structure of SARS‐CoV‐2 does not rule out a laboratory origin“
mitgearbeitet haben könnte sich zu einem echten „Game Changer“ entwickeln. Immerhin erachten die USA die Arbeit der Forscher um Rosana Segreto für derart zentral, daß sie am 15.1. auf der Webseite der Regierung die durch ihre Forschungen aufgeworfenen Fragen als eine der drei Fragen hervorhebt, die Inspekteure der WHO zu beantworten hätten:
Die Bidon-Regierung hat nach ihrem Amtsantritt diesen Hinweis wieder von der Regierugsseite genommen.
In ihrer Arbeit legen die Forscher um Rosana Segreto nämlich dar, daß es wissenschaftlich nicht haltbar ist, daß Covid-19 nur auf natürlichem Weg entstanden sein kann. Damit versetzen sie die Anhänger der Theorie eines natürlichen Ursprungs in argumentativen Zugzwang.
Doch nicht nur das, sie vertreten darüber hinaus auch die Auffassung, daß
„SARS‐COV‐2 chimeric structure and furin cleavage site might be the result of genetic manipulation„,
also daß mindestens Teile von Covid-19 aus dem Labor stammen.
Doch – wohl ohne es zu wissen – bestätigen die Forscher damit zugleich Aussagen von hochrangigen Persönlichkeiten, wie z.B. dem Präsident der „Weltakademie für Biotechnologie (WABT)“ Prof. Joseph Tritto aus dem August 2020, die wir hier bereits ausführlich vorgestellt haben
Wer die Frage nach dem Ursprung von Covid-19 stellt, stellt auch die Schuldfrage
Das Covid-19-Virus – genauer gesagt müßte man sagen „Die Maßnahmen der Regierungen zur Zurückdrängung von Covid-19″ – hat nach Einschätzung des weltgrößten Rückversicherers, der „Münchner Rück“ einen Schaden von 12.000 Milliarden Euro verursacht. Die damit eigentlich zwangsläufig verbundene Frage, wie viele davon der Verursacher bezahlt wird erst gar nicht gesellt. Schon gar nicht in Richtung China, wo das Virus zumindest unstreitig seinen geographischen Ursprung hat.
Was wäre aber, wenn Covid-19 eine menschengemachte Konstruktion aus einem Labor in China wäre? Was wäre, wenn diese „Chimäre“ aus Versehen aus dem Labor entwichen wäre, oder was wäre wenn diese „Chimäre“ absichtlich aus dem Labor entlassen worden wäre? Wie würde sich die Frage der Verantwortung für die Schäden dann stellen?
Zweifel an den offiziellen Erklärungen gab es von Anbeginn an, wie dieser Beitrag der alternativen Medien von Ende April 2020 belegt:
Fragen über Fragen, die bisher niemand stellt
Tatsache ist daß es seit etwa 20 Jahren Forschungen an SATS-Chimären gibt. Tatsache eist, daß das Wuhan Institute of Virology (WIV) an diesen Forschungen beteiligt war. Tatsache ist, daß das Wuhan Institute of Virology (WIV) an dem Virus RaTG13 gearbeitet hat, der eine 100% „nucleotide identity“ mit der Sequenz „BtCoV/4991“ aufwiest, die das WIV aus einer Höhle in Yunnan entnommen hat. Tatsache ist, daß die Arbeiten an RaTG13 dadurch verschleiert wurden, daß sie mit Hilfe eines Doppeleintrags in eine Datenbank eingegeben wurden. Tatsache ist, daß die Datenbank des WIV “Wildlife‐borne viral pathogen database” in “Bat and rodent‐borne viral pathogen database,” umbenannt wurde und in ihrer Beschreibung der Begriff “wild animal” in “bat and rodent” geändert wurde und daß die Erwähnung von “arthropod vectors” gelöscht wurden. Tatsache ist, daß die gesamte Datenbank seit Anfang Mai gelöscht wurde. Tatsache ist außerdem, daß zur Erzeugung des Covid-19-Virus eine Fledermaus aus Yunnan im 1500Km entfernteren Guangdong mit einem dort gar nicht vorkommendes und auch sost. vom Aussterben bedrohtes malaysisches Gürteltier hätte irgendwie so interagieren müssen, daß zum selben Zeitpunkt das Gürteltier-SATS und das Fledermaus-SARS die selbe Zelle hätten befallen müssen.
All diese Fragen und noch viel mehr Fragen müssten eigentlich beantwortet werden, wen man ernsthaft die Behauptung vertritt daß Covid-19 rein natürlichen Ursprungs wäre. Statt diese Fragen zu beantworten, schweigen die Anhänger der Theorie eines natürlichen Ursprungs lieber und bezeichnen die Zweifler an dieser Theorie als „Verschwörungstheoretiker“ und machen vor allem eines nicht: Sie fördern bisher keinerlei Forschung dahingehend, daß die Frage nach einem nicht natürlichen Ursprung beantwortet wird!
Hinweise auf künstlichen Ursprung
In einem Interview mit der Austrian Press Agency (APA) legt Frau Segreto auch ihre Argumente gegen einen natürlichen Ursprung des Covid-19-irus offen:
Desinteresse bei Offiziellen alle Fragen zu beantworten
Erstaunlich ist auch das Verhalten vieler sonstiger Vertreter der Behörden, Forschung etc. Vielfach ducken sie sich weg, schweigen und/oder versuchen Fragesteller in die Irre zu führen.
Dies beginnt schon beim Labor in Wuhan. Bei einem Schaden von 12.000 Milliarden Euro weltweit dürfte man erwarten, daß man sich dieses Labor einmal genauer ansieht. Foch weit gefehlt:
Dies deckt sich mit dem Buch von Prof Tritto, in dem er von den Ursprüngen on Covid-19 berichte. Niemand kennt es und er bleibt trotz seiner Reputation und Aussagen unbeachtet
Dann fand Frau Segreto zunächst kein Medium für die Veröffentlichung ihrer wissenschaftlichen Arbeit:
Auch die Anfrage der AfD an die Staatsregierung zu diesem Thema zeigt ein identisches Verhalten. Zwar muss die Staatsregierung Fragen der Opposition beantworten, doch die Qualität der Antworten ist erschreckend und von Wegducken, Leugnen, und Indieirreführen gekennzeichnet. Anschaulich nachvollziehbar in der Antwort der Bayerischen Staatsregierung in Drucksache 18/10909 auf eine entsprechende Anfrage der AfD im Landtag.
Dies ergibt sich schon daraus, dass die Staatsregierung im LGL Professoren und promovierte Wissenschaftler beschäftigt, denen die Kenntnis eines kompletten Fachgebiets zuzurechnen ist, und zwar völlig unabhängig von der Frage ihres tatsächlichen Kenntnisstands.
Im Lichte der Arbeit von Rossana Segreto Yuri Deigin, 17.11.2020 erscheint die einen Monat später unter 3.1. der Drucksache 18/10909 gegebene Antwort
unzutreffend. Außerdem dürfte dieser Hinweis irreführend sein, so weit damit intendiert ist, im Umkehrschluss das Bild aufzubauen, dass es wissenschaftlich belegt sei, dass Cobd-19 auf natürlichem Weg entstanden sei. Wenn die Staatsregierung außerdem argumentiert,
scheint sie beim Leser das irrige Bild erzeigen zu wollen, dass Wissenschaft eine demokratische Einrichtung sei, ganz so, als ob man drüber abstimmen könnte, ob die Erde eine Scheibe ist, oder eine Kugel.
Die genetische Struktur von SARS-CoV-2 schließt einen Labor-Ursprung nicht aus
In Folge übersetzen wir die Arbeit des Forscherteams aus Innsbruck exklusiv und vollständig für unsere Leser. Die Arbeit ist als „Creative Commons“ lizensiert und damit für jedermann nutzbar. Selbstverständlich verwiesen wir gerne auf die Quelle und ergänzen, daß der Text nach den Aufzählungspunkten ausschließlich durch die Autoren verfaßt wurde.
Damit der Leser die Möglichkeit hat auch die Übersetzung zu kontrollieren, führen wir zuerst den englischen Originaltext auf und dann die zugehörige Übersetzung.
Abstract / Zusammenfassung
- Severe acute respiratory syndrome‐coronavirus (SARS‐CoV)‐2′s origin is still controversial. Genomic analyses show SARS‐CoV‐2 likely to be chimeric, most of its sequence closest to bat CoV RaTG13, whereas its receptor binding domain (RBD) is almost identical to that of a pangolin CoV. Chimeric viruses can arise via natural recombination or human intervention. The furin cleavage site in the spike protein of SARS‐CoV‐2 confers to the virus the ability to cross species and tissue barriers, but was previously unseen in other SARS‐like CoVs. Might genetic manipulations have been performed in order to evaluate pangolins as possible intermediate hosts for bat‐derived CoVs that were originally unable to bind to human receptors? Both cleavage site and specific RBD could result from site‐directed mutagenesis, a procedure that does not leave a trace. Considering the devastating impact of SARS‐CoV‐2 and importance of preventing future pandemics, researchers have a responsibility to carry out a thorough analysis of all possible SARS‐CoV‐2 origins.
- Der Ursprung des schweren Coronavirus (SARS-CoV) 2 mit akutem respiratorischem Syndrom ist immer noch umstritten. Genomanalysen zeigen, dass SARS-CoV-2 wahrscheinlich chimär ist, wobei der größte Teil seiner Sequenz dem Fledermaus-CoV-RaTG13 am nächsten kommt, während seine Rezeptorbindungsdomäne (RBD) fast identisch mit der eines Pangolin-CoV ist. Chimäre Viren können durch natürliche Rekombination oder menschliches Eingreifen entstehen. Die Furin-Spaltstelle im Spike-Protein von SARS-CoV-2 verleiht dem Virus die Fähigkeit, Spezies und Gewebebarriere zu überwinden, war aber bei anderen SARS-ähnlichen CoVs bisher nicht sichtbar. Könnten genetische Manipulationen durchgeführt worden sein, um Pangoline als mögliche Zwischenwirte für von Fledermäusen abgeleitete CoVs zu bewerten, die ursprünglich nicht an menschliche Rezeptoren binden konnten? Sowohl die Spaltstelle als auch die spezifische RBD könnten aus einer ortsgerichteten Mutagenese resultieren, einem Verfahren, das keine Spuren hinterlässt. Angesichts der verheerenden Auswirkungen von SARS-CoV-2 und der Bedeutung der Verhinderung künftiger Pandemien sind die Forscher dafür verantwortlich, eine gründliche Analyse aller möglichen SARS-CoV-2-Ursachen durchzuführen.
INTRODUCTION / Einleitung
- Nearly a year has passed since the outbreak of severe acute respiratory syndrome‐coronavirus 2 (SARS‐CoV‐2) in Wuhan, China, and its origin is still controversial. Despite the international research effort conducted, a natural host, either direct or intermediate, has not yet been identified. The hypothesis that the Wuhan Huanan Seafood Wholesale Market was the first source for animal–human virus transmission has now been conclusively dismissedi and the few market samples that were collected showed only human‐adapted SARS‐CoV‐2, with no traces of zoonotic predecessor strainsii . Almost all scientific papers published to date purport that SARS‐CoV‐2 has a natural origin, and the only published paper considering possible a lab origin[1] focuses on serial passage as the technique that could justify SARS‐CoV‐2 special adaptation to human cells. We here describe how the two main SARS‐CoV‐2 features, (1) the presence of a furin cleavage site missing in other CoVs of the same group and (2) an receptor binding domain (RBD) optimized to bind to human cells[2] might be the result of lab manipulation techniques such as site‐directed mutagenesis. The acquisition of both unique features by SARS‐CoV‐2 more or less simultaneously is less likely to be natural or caused only by cell/animal serial passage.
- Fast ein Jahr ist seit dem Ausbruch des schweren akuten respiratorischen Syndroms Coronavirus 2 (SARS-CoV-2) in Wuhan, China, nun vergangen, und sein Ursprung ist immer noch umstritten. Trotz der internationalen Forschungsanstrengungen konnte ein natürlicher Wirt, weder direkt, noch indirekt identifiziert werden. Die Hypothese, dass der Großhandelsmarkt für Meeresfrüchte in Wuhan Huanan die erste Quelle für die Übertragung von Tier-Mensch-Viren war, wurde inzwischen endgültig verworfen, denn die wenigen überhaupt auf diesem Markt gesammelten Proben zeigten nur menschlich angepasstes SARS-CoV-2 ohne Spuren von zoonotischen Vorgängerstämmen aufzuweisen. Fast alle bisher veröffentlichten wissenschaftlichen Arbeiten geben dennoch an, dass SARS-CoV-2 einen natürlichen Ursprung hat, und die einzige veröffentlichte Arbeit, die einen möglichen Laborursprung in Betracht zieht [1], konzentriert sich auf eine „serielle Passage“ als einer Technik, die eine spezielle Anpassung von SARS-CoV-2 an menschliche Zellen rechtfertigen könnte. Im nun folgenden Beitrag beschreiben wir, wie die beiden Hauptmerkmale von SARS-CoV-2: (1) das Vorhandensein einer Furin-Spaltstelle, die in anderen CoVs derselben Gruppe fehlt, und (2) eine Rezeptorbindungsdomäne (RBD), die für die Bindung an menschliche Zellen optimiert ist [ 2] könnte ein Ergebnis von Labormanipulationstechniken wie einer ortsgerichteten Mutagenese sein. Es ist weniger wahrscheinlich, dass der gleichzeitige Erwerb beider beiden einzigartigen Merkmale durch SARS-CoV-2 zu einem mehr oder weniger selben Zeitpunkt natürlichen Ursprungs ist, als daß er ausschließlich durch eine serielle Passage über Zellen und Tiere erreicht wurde.
SARS‐COV‐2′S CLOSEST RELATIVES ARE BAT AND PANGOLIN CORONAVIRUSES
Die engsten Verwandten von SARS-COV-2 sind Fledermaus- und PANGOLIN-CORONAVIREN
Inzwischen ist allgemein bekannt, daß Fledermaus- und Gürteltier-Coronaviren diejenigen sind, die Covid-19 am ähnlichsten sind. Doch so allgemein ausgedrückt ist das irreführend. Genauer gesagt, handelt es sich beim Fledermausvirus um RaTG13 MN996532, einem alten Beamten der Forscher aus dem WIV:
- Zhou et al.[3] from the Wuhan Institute of Virology (WIV) were the first to identify and characterize a new coronavirus (CoV), SARS‐CoV‐2. The genomic sequences obtained from early cases shared 79% sequence identity to the CoVs that caused severe acute respiratory syndrome (SARS‐CoV) in 2002–2003 and 96.2% sequence identity to RaTG13 (MN996532), a CoV sequence detected from a Rhinolophus affinis bat. RaTG13 is currently the closest phylogenetic relative for SARS‐CoV‐2 found,[4] but its complete genomic sequence was not published before the outbreak of SARS‐CoV‐2 and the original sample was collected in the Yunnan province (China) by the same group of WIV researchers in 2013. Zhou et al.[3] stated to have found a match between SARS‐CoV‐2 and a short region of RNA‐dependent RNA polymerase (RdRp) of a CoV in their database and then fully sequenced the original sample collected in 2013, which they called RaTG13.
- Zhou et al. [3] vom Wuhan Institute of Virology (WIV) identifizierten und charakterisierten als erste ein neues Coronavirus (CoV), SARS-CoV-2. Die aus frühen Fällen erhaltenen Genomsequenzen hatten eine Sequenzidentität von 79% mit den CoVs, die 2002–2003 ein schweres akutes respiratorisches Syndrom (SARS-CoV) verursachten, und eine Sequenzidentität von 96,2% mit RaTG13 (MN996532), einer CoV-Sequenz, die aus einer Fledermaus von Rhinolophus affinis nachgewiesen wurde. RaTG13 ist derzeit der engste phylogenetische Verwandte für SARS-CoV-2, [4] aber seine vollständige Genomsequenz wurde nicht vor dem Ausbruch von SARS-CoV-2 veröffentlicht worden und außerdem wurde die Originalprobe in der Provinz Yunnan (China) von der selben Gruppe von WIV-Forschern gesammelt, wie im Jahr 2013. Zhou et al. [3] gaben an, eine Übereinstimmung zwischen SARS-CoV-2 und einer kurzen Region der RNA-abhängigen RNA-Polymerase (RdRp) eines CoV in ihrer Datenbank gefunden zu haben, und sequenzierten dann die 2013 gesammelte Originalprobe, die sie RaTG13 nannten, vollständig.
Pikant ist auf jeden Fall, daß die bis dahin unbekannte Variante RaTG13, an der das WIV Experimente durchgeführt hat, – aus welchen Gründen auch immer – auf eine solche Art und Weise in den Datenbanken eingetragen war, daß man sie bisher nicht finden und damit auch nicht untersuchen konnte:
- We discovered that the RdRp of RaTG13 has 100% nucleotide identity with the sequence BtCoV/4991 (KP876546), which was identified by Ge et al.[5] in a Rhinolophus affinis bat in the Yunnan province in 2013, same location and year as RaTG13. BtCoV/4991 was collected in a mine colonized by bats near Tongguanzhen, Mojiang, Yunnan. The WIV researchers were invited to investigate the mine after six miners there had contracted severe pneumonia in 2012iii , and three of the miners have died.[6] The miners have been tasked with clearing out bat droppings in the mine, and the severity of their pneumonia correlated with the duration of exposure to the mine.[7] Four miners’ samples subsequently underwent testing at WIV, where Immunoglobulin G (IgG) antibodies against SARS were identified in all samples.[8] Considering that only about 5300 people were infected in mainland China during the SARS outbreak of 2002–2004, most of whom resided in Guandong, the odds of four miners in Yunnan retaining antibodies from the 2002–2004 SARS outbreak are negligible. On the other hand, it is possible that the SARS antibody test administered to the miners cross‐reacted with a novel SARS‐like bat virus that the miners had acquired at the mine. Ge et al.[5] have identified a number of CoVs in the mine, but based on the phylogenetic analysis, BtCoV/4991 was the only SARS‐related strain, clearly separated from all known alpha‐ and beta‐CoVs at that time. BtCoV/4991 was also different from other bat CoVs in the phylogenetic analysis carried out by Wang et al. in 2019.[9] Chen et al.[10] identified BtCoV/4991 as the closest sequence to SARS‐CoV‐2 because RaTG13 had not yet been published at that time. BtCoV/4991 and RaTG13 have been later asserted to be two different coding names of the same strain, as their original authors at WIV registered the two strains as one entry in the Database of Bat‐associated Viruses (DBatVir).iv
- Wir entdeckten, dass der RdRp von RaTG13 eine 100% ige Nucleotididentität mit der Sequenz BtCoV / 4991 (KP876546) aufweist, die von Ge et al. [5] in einer Fledermaus von Rhinolophus affinis in der Provinz Yunnan im Jahr 2013 Identifiziert wurde, also dem selben Ort und Jahr wie RaTG13. BtCoV / 4991 wurde wiederum in einer von Fledermäusen besiedelten Mine in der Nähe von Tongguanzhen, Mojiang, Yunnan gesammelt. Die WIV-Forscher wurden eingeladen, die Mine zu untersuchen, nachdem sich 2012 sechs Bergleute dort eine schwere Lungenentzündung zugezogen hatten, an der drei der Bergleute gestorben sind. Die Bergleute wurden beauftragt, Fledermauskot in der Mine zu beseitigen, und die Schwere ihrer Lungenentzündung korrelierte mit der Dauer der Exposition gegenüber der Mine. Vier Minenarbeiterproben wurden anschließend bei WIV getestet, wo in allen Proben Immunglobulin G (IgG) -Antikörper gegen SARS identifiziert wurden. In Anbetracht der Tatsache, dass während des SARS-Ausbruchs von 2002 bis 2004 nur etwa 5300 Menschen auf dem chinesischen Festland infiziert waren, von denen die meisten in Guandong lebten, ist die Wahrscheinlichkeit, dass vier Bergleute in Yunnan Antikörper aus dem SARS-Ausbruch von 2002 bis 2004 zurückhalten, vernachlässigbar. Andererseits ist es möglich, dass der den Bergleuten verabreichte SARS-Antikörpertest mit einem neuartigen SARS-ähnlichen Fledermausvirus kreuzreagierte, das die Bergleute in der Mine erworben hatten. Ge et al. [5] haben eine Reihe von CoVs in der Mine identifiziert, aber basierend auf der phylogenetischen Analyse war BtCoV / 4991 der einzige SARS-verwandte Stamm, der zu diesem Zeitpunkt klar von allen bekannten Alpha- und Beta-CoVs getrennt war. BtCoV / 4991 unterschied sich auch von anderen Fledermaus-CoVs in der phylogenetischen Analyse von Wang et al. im Jahr 2019. [9] Chen et al. [10] BtCoV identifiziert 4991 nur deswegen als die SARS-CoV-2 am nächsten liegende Sequenz, da RaTG13 zu jenem Zeitpunkt noch nicht veröffentlicht worden war. Es wurde dann später behauptet, dass BtCoV / 4991 und RaTG13 zwei verschiedene Codierungsnamen desselben Stammes sein sollen, da ihre ursprünglichen Autoren bei WIV die beiden Stämme als einen gemeinsamen Eintrag in der Datenbank der Fledermaus-assoziierten Viren (DBatVir) .iv eingetragen haben
Im Juli 2020 haben die chinesischen Forscher des Wuhan-Instituts (WIV) dann zugegeben, daß sie an der RaTG13-Probe, als der Probe, die Covid-19 am ähnlichsten ist, bereits im Jahr 2018 Experimente durchgeführt haben, was praktisch bedeutet, daß im Wuhan-Institut, also im Labor bereits im Jahr 2018 an dem Virus gearbeitet wurde, das dann dem am ähnlichsten ist, das Ende 2019 im Seafood-Market erstmals Personen anstecken wird.
- In late July 2020, Zhengli Shi, the leading CoV researcher from WIV, in an email interview [11] asserted the renaming of the RaTG13 sample and unexpectedly declared that the full sequencing of RaTG13 has been carried out as far back as in 2018 and not after the SARS‐CoV‐2 outbreak, as stated in Zhou et al.[3] The reversal in WIV’s stance on when exactly RaTG13 was fully sequenced could have been due to the discovery by independent researchers into the origins of SARS‐CoV‐2 that the filenames of the raw sequencing reads deposited by WIV on May 19, 2020v seem to indicate that sequencing for RaTG13 was done in 2017 and 2018.vi However, no formal erratum about year of sequencing and sample renaming from the authors of Zhou et al. [3] has yet appeared, or as far as is currently known, has been submitted.
- Erst Ende Juli 2020 bestätigte Zhengli Shi, die führende CoV-Forscher von WIV, in einem E-Mail-Interview [11] die Umbenennung der RaTG13-Probe und gestand überraschend zu, dass die vollständige Sequenzierung von RaTG13 bereits im Jahr 2018 durchgeführt worden war und nicht erst nach dem SARS-CoV-2-Ausbruch, wie in Zhou et al. [3] behauptet hatten. Diese Kehrtwende in der Argumentation von WIV, wann genau RaTG13 vollständig sequenziert wurde, könnte auf die Entdeckung unabhängiger Forscher zu den Ursprüngen von SARS-CoV-2 zurückzuführen sein, denen gemäß die Dateinamen der von WIV am 19. Mai 2020v hinterlegten rohen Sequenzierungslesungen darauf hinzudeuten scheinen, daß diese Sequenzierung für RaTG13 bereits in den Jahren 2017 und 2018 durchgeführt wurde. vi Die Autoren Zhou et al. [3] haben auf diesen Fehler jedoch nicht durch ein formelles Erratum über das Jahr der Sequenzierung und Probenumbenennung aufmerksam gemacht noch ist in solcher Hinweis sonstwo erschienen oder wurde anderweitig bekannt gemacht, oder eingereicht.
Ein weiteres nichtmenschliches Element von Covid-19 stammt von einem „Malaysian Pangolin„. Dieses Element ist wiederum für den Eintritt in den Körper, für die Replikation und für die Pathogenese zuständig. Er ist gemäß der Forschungen von Frau Segreto aber die einzige Variante, die für den Menschen ansteckend ist. Die der Fledermaus alleine ist jedenfalls nicht für den Menschen ansteckend. Dieser „Malaysian Pangolin„, den man also benötigt, um eine Ansteckung beim Menschen zu erreichen, ist in China jedoch weitgehend ausgerottet und wird aus den südlich an China angrenzenden Ländern illegal importiert.
Doch besteht auf die Populationen des Malaiischen Schuppentiers noch immer ein extrem großer Druck durch Jagd. Seit dem Inkrafttreten des CITES-Abkommens ist der Bestand dramatisch gesunken. Neben der Jagd hat auch der Lebensraumverlust infolge der Abholzung der Wälder großen Einfluss auf die Bestände. Zwar kann das Malaiische Schuppentier auch in von Menschen genutzten Gebieten überleben, doch sind dichte Wälder mit großen Bäumen und natürlichen Unterschlupfmöglichkeiten zur Aufzucht des Nachwuchses offensichtlich notwendig. All dies führte dazu, dass in den nördlichen Regionen des Verbreitungsgebietes, so etwa in Laos und Thailand, die Populationen des Malaiischen Schuppentiers nahezu zusammengebrochen sind,[24] die extensive Jagd hat dabei in den letzten Jahren die südlichen Verbreitungsgebiete erreicht.[11] Untergeordnet kommen vereinzelt Tiere auch bei Verkehrsunfällen zu Tode.[25][1] Die IUCN geht insgesamt von einem Rückgang der Populationen seit den 1990er Jahren um nahezu 80 % aus. Daher stufte sie die Art 2014 von der Gefährdungskategorie „stark gefährdet“ (endangered) auf „vom Aussterben bedroht“ (critically endangered) hoch. Die Schuppentierart ist in mehreren geschützten Gebieten innerhalb des Verbreitungsgebietes präsent
- The second non‐human RdRp sequence closest to BtCoV/4991 (91.89% nucleotide identity) is the CoV sequence MP789 (MT084071) isolated in 2019 in a Malaysian pangolin (Manis javanica) from the Guangdong province (GD), China.[12] The envelope protein of MP789 shows surprisingly 100% aminoacidic identity with the corresponding protein in RaTG13, in bat‐SL‐CoVZXC21 (MG772934.1), in bat‐SL‐CoVZC45 (MG772933.1) and in some early SARS‐CoV‐2 isolates (e.g. YP_009724392).[13] The envelope protein of CoVs is involved in critical aspects of the viral lifecycle, such as viral entry, replication and pathogenesis.[14]
- Die zweite nicht-humane RdRp-Sequenz, die BtCoV / 4991 am nächsten kommt (91,89% Nucleotididentität), ist die CoV-Sequenz MP789 (MT084071), die 2019 in einem malaysischen Pangolin (Manis javanica) aus der Provinz Guangdong (GD), China, isoliert wurde. [12] Das Hüllprotein von MP789 zeigt eine überraschend 100% ige Aminosäureidentität mit dem entsprechenden Protein in RaTG13, in Fledermaus-SL-CoVZXC21 (MG772934.1), in Fledermaus-SL-CoVZC45 (MG772933.1) und in einigen frühen SARS-CoV-2 Isolate (zB YP_009724392). [13] Das Hüllprotein von CoVs ist an kritischen Aspekten des viralen Lebenszyklus beteiligt, wie z. B. dem Eintritt, der Replikation und der Pathogenese von Viren. [14]
Um aber ein Fledermaus-SARS-Virus mit SARS-Viren des Pangolin auf natürlichem Weg zu vereinen, müßten sich beide Tiere nicht nur treffen, sondern auch noch ihre Viren austauschen.
.
BAT COVS HAVE BEEN THOROUGHLY STUDIED AND GENETICALLY MANIPULATED
Covid-Viren von Fledermäusen wurden bereits früher gründlich untersucht und genetisch manipuliert
Es gab nämlich bereits Forschungen chimäre CoVs zu entwickeln, die aus Fledermaus-CoV-Grundgerüsten bestehen und die normalerweise keine menschlichen Zellen infizieren können und deren Spike-Proteine durch solche aus CoVs mit menschlichem ACE2 ersetzt wurden.
Diese Chimären sollten eigentlich Rekombinationsereignisse simulieren, die in der Natur auftreten könnten. [19, 20] Solche Experimente zum Funktionsgewinn haben aber eine Reihe von Bedenken hinsichtlich der biologischen Sicherheit aufgeworfen und bei Forschern und der Öffentlichkeit Kontroversen ausgelöst. Eines der Hauptargumente zugunsten von Funktionsgewinnstudien ist die Notwendigkeit, mit einem Arsenal an Medikamenten und Impfstoffen auf die nächste Pandemie vorbereitet zu sein. Im Gegensatz dazu ist eines der Hauptargumente gegen sie, dass die nächste Pandemie durch diese Experimente aufgrund des Risikos einer Laborflucht verursacht werden könnte.
- Many studies have reported that bats are natural reservoirs for a broad diversity of potentially pathogenic SARS‐like CoVs.[15, 16] Some of these viruses can potentially directly infect humans[17], whereas others need to mutate their spike protein in order to effectively bind to the human angiotensin 1‐converting enzyme 2 (hACE2) receptor and mediate virus entry.[18] In order to evaluate the emergence potential of novel CoVs, researchers have created a number of chimeric CoVs, consisting of bat CoV backbones, normally unable to infect human cells, whose spike proteins were replaced by those from CoVs compatible with human ACE2. These chimeras were meant to simulate recombination events that might occur in nature.[19, 20] Such gain‐of‐function experiments have raised a number of biosafety concerns and stirred controversy among researchers and the general public. One of the main arguments in favor of gain‐of‐function studies is the need to be prepared with an arsenal of drugs and vaccines for the next pandemic.[21] By contrast, one of the main arguments against them is that the next pandemic itself could be caused by those experiments, due to the risk of lab escape.[22, 23]In recent years, the field of corona‐virology had been focused on pan‐CoV therapies and vaccines, as evident from research conducted in the past 5 years,[24-27] as well as from media reports.vii Synthetically generating diverse panels of potential pre‐emergent CoVs was declared a goal of active grants for the EcoHealth Alliance, which funded some of such research at WIV, in collaboration with laboratories in the USA and other international partners.viii
- In zahlreichen Studien wurde bereits dargelegt, dass Fledermäuse natürliche Reservoire für eine breite Vielfalt potenziell pathogener SARS-ähnlicher CoVs sind. [15, 16] Einige dieser Viren können den Menschen möglicherweise direkt infizieren [17], während andere ihr Spike-Protein mutieren müssen, um dies zu tun effektiv an den humanen Angiotensin 1-konvertierenden Enzym 2 (hACE2) -Rezeptor binden und den Viruseintritt vermitteln. [18] Um das Entstehungspotenzial neuartiger CoVs zu bewerten, haben Forscher eine Reihe chimärer CoVs entwickelt, die aus Fledermaus-CoV-Grundgerüsten bestehen, die normalerweise keine menschlichen Zellen infizieren können und deren Spike-Proteine durch solche aus CoVs mit menschlichem ACE2 ersetzt wurden. Diese Chimären sollten Rekombinationsereignisse simulieren, die in der Natur auftreten könnten. [19, 20] Solche Experimente zum Funktionsgewinn haben eine Reihe von Bedenken hinsichtlich der biologischen Sicherheit aufgeworfen und bei Forschern und der Öffentlichkeit Kontroversen ausgelöst. Eines der Hauptargumente zugunsten von Funktionsgewinnstudien ist die Notwendigkeit, mit einem Arsenal an Medikamenten und Impfstoffen auf die nächste Pandemie vorbereitet zu sein. Im Gegensatz dazu ist eines der Hauptargumente gegen sie, dass die nächste Pandemie durch diese Experimente aufgrund des Risikos einer Laborflucht verursacht werden könnte. In den letzten Jahren konzentrierte sich das Gebiet der Koronavirologie auf Pan-CoV-Therapien und -Impfstoffe, wie aus den in den letzten 5 Jahren durchgeführten Forschungsarbeiten [24-27] sowie aus Medienberichten hervorgeht.vii Synthetische Erzeugung verschiedener Panels von Potenzielle CoVs vor dem Auftauchen wurden zum Ziel aktiver Zuschüsse für die EcoHealth Alliance erklärt, die in Zusammenarbeit mit Laboratorien in den USA und anderen internationalen Partnern einen Teil dieser Forschung bei WIV finanzierte.
CREATING CHIMERIC COVS WITH NOVEL RBDS HAS GONE ON FOR DECADES
Das Erstellen von Covid-Chimären mit neuartigen RBDS gibt es bereits seit Jahrzehnten
- Researchers have been generating chimeric CoVs for over two decades, long before the advent of modern sequencing or genetic engineering techniques. For example, in 1999, a group from Utrecht University used targeted RNA recombination to create a “cat‐and‐mouse” CoV chimera: the RBDs of a feline and murine CoV were swapped, demonstrating that this exchange swapped also species tropism during in vitro experiments.[28]
- Forscher erzeugen seit über zwei Jahrzehnten chimäre CoVs, lange bevor moderne Sequenzierungs- oder Gentechnik-Techniken aufkamen. Beispielsweise verwendete eine Gruppe der Universität Utrecht 1999 eine gezielte RNA-Rekombination, um eine „Cat-and-Mouse“ -CoV-Chimäre zu erzeugen: Die RBDs eines Katzen- und Maus-CoV wurden ausgetauscht, was zeigt, dass dieser Austausch auch den Tropismus von Arten in vitro austauschte Experimente. [28]
2007: Die Forschergruppe um Frau Shi schuf Pseudoviren mit humanen Immundefizienzviren (HIV) mit Fledermaus-CoV-Spike-Proteinen
- In 2007, the Shi group at WIV created a series of “bat‐man” CoV chimeric spike proteins while trying to determine what exactly confers CoVs the ability to jump from one species to another. The researchers used different segments of the spike protein of the human SARS virus to replace corresponding segments in the spike protein of a bat viral backbone. It was concluded that a relatively short region (aa 310 to 518) of the spike protein “was necessary and sufficient to convert Rp3‐S into a huACE2‐binding molecule,”29 that is to provide the bat CoV spike protein with a novel ability of binding to a human ACE2 receptor.
- Bereits 2007 schuf die Shi-Gruppe am WIV eine Reihe von chimären „Bat-Man“-CoV-Spike-Proteinen, um herauszufinden, was CoVs genau die Fähigkeit verleiht, von einer Spezies zur anderen überzuspringen. Die Forscher verwendeten verschiedene Segmente des Spike-Proteins des humanen SARS-Virus, um entsprechende Segmente im Spike-Protein eines Fledermaus-Virus-Rückgrats zu ersetzen. Es wurde daraus der Schluss gezogen, dass eine relativ kurze Region (aa 310 bis 518) des Spike-Proteins „notwendig und ausreichend ist, um Rp3-S in ein huACE2-bindendes Molekül umzuwandeln“ 29, um dem CoV-Spike-Protein der Fledermaus eine neue Fähigkeit zu verleihen der Bindung an einen menschlichen ACE2-Rezeptor.
2008: Die Forschergruppe um Baric schuf eine lebende Covid-Chimäre
- In 2008, the Baric group at the University of North Carolina (UNC) took the WIV research one step further: instead of using human immunodeficiency viruses (HIV) pseudo‐viruses with bat CoV spike proteins, a live chimeric CoV was created. Following the experiments of their 2007 WIV colleagues, the Baric group used a bat SARS‐like CoV as a backbone and replaced its RBD with the RBD from human SARS.[30]
- Im Jahr 2008 ging die Baric-Gruppe an der University of North Carolina (UNC) einen Schritt weiter mit der WIV-Forschung: Anstatt Pseudoviren mit humanen Immundefizienzviren (HIV) mit Fledermaus-CoV-Spike-Proteinen zu verwenden, wurde ein lebendes chimäres CoV erstellt. Nach den Experimenten ihrer WIV-Kollegen von 2007 verwendete die Baric-Gruppe ein Fledermaus-SARS-ähnliches CoV als Rückgrat und ersetzte seine RBD durch die RBD aus menschlichem SARS. [30]
2015: Die Forschergruppe um Shi und Baric schufen gemeinsam eine weitere lebende Covid-Chimäre
- In 2015, the Shi and Baric groups joined forces and published probably the most famous gain‐of‐function virology paper, which described the creation of another synthetic chimeric virus.[19] This time the RBD of a mouse‐adapted SARS backbone (SARS‐MA15) was replaced by the RBD of RsSHC014, a bat strain previously isolated from Yunnan bats in 2011 by the Shi group. In 2016, the Baric group repeated their 2015 experiment using the same SARS‐MA15 backbone and the RBD from Rs3367,[31] a close relative of RsSHC014 also previously found in Yunnan by WIV and renamed “WIV1” after live culturing.[17]
- Im Jahr 2015 schlossen sich die Shi- und Baric-Gruppen zusammen und veröffentlichten das wahrscheinlich berühmteste Virologiepapier mit Funktionsgewinn, in dem die Entstehung eines weiteren synthetischen chimären Virus beschrieben wurde. [19] Diesmal wurde die RBD eines an die Maus angepassten SARS-Rückgrats (SARS-MA15) durch die RBD von RsSHC014 ersetzt, einem Fledermausstamm, der 2011 von der Shi-Gruppe aus Yunnan-Fledermäusen isoliert worden war. 2016 wiederholte die Baric-Gruppe ihr Experiment von 2015 unter Verwendung des gleichen SARS-MA15-Rückgrats und der RBD von Rs3367 [31], einem nahen Verwandten von RsSHC014, der ebenfalls zuvor von WIV in Yunnan gefunden und nach Lebendkultivierung in „WIV1“ umbenannt wurde. [17]
2017: Die Shi-Gruppe arbeitete an weiteren Chimären
- Probably the largest reported number of novel chimeric viruses created was described in a 2017 paper from the Shi group at WIV,[15] in which the authors reported creating eight chimeric viruses using WIV1 as a backbone and transplanting into it various RBDs from bat SARS‐like viruses. These viruses were collected over a span of 5 years from the same cave near Kunming, Yunnan Province, where the Shi group originally found Rs3367 and RsSHC014. Only two of the eight live chimeric viruses were successfully rescued, and those two strains were found to possess the ability to bind to the human ACE2 receptor, as confirmed by experiments in hACE2‐expressing HeLa cells and RT‐PCR quantification of viral RNA.
- Die wahrscheinlich größte gemeldete Anzahl neuartiger chimärer Viren wurde in einem Artikel der Shi-Gruppe von WIV aus dem Jahr 2017 beschrieben [15], in dem die Autoren berichteten, acht chimäre Viren mit WIV1 als Rückgrat zu erzeugen und verschiedene RBDs aus Fledermaus-SARS in dieses zu transplantieren wie Viren. Diese Viren wurden über einen Zeitraum von 5 Jahren in derselben Höhle in der Nähe von Kunming, Provinz Yunnan, gesammelt, wo die Shi-Gruppe ursprünglich Rs3367 und RsSHC014 gefunden hatte. Nur zwei der acht lebenden chimären Viren wurden erfolgreich gerettet, und es wurde festgestellt, dass diese beiden Stämme die Fähigkeit besitzen, an den menschlichen ACE2-Rezeptor zu binden, was durch Experimente in hACE2-exprimierenden HeLa-Zellen und RT-PCR-Quantifizierung von viraler RNA bestätigt wurde.
.
SARS‐COV‐2 SHARES ITS RBD WITH A PANGOLIN COV
SARS-COV-2 TEILT SEINE RBD MIT EINEM PANGOLIN-COV
- The possibility that pangolins could be the intermediate host for SARS‐CoV‐2 has long been under discussion. [32-34] The biggest divergence between SARS‐CoV‐2 and RaTG13 is observed in the RBD of their spike proteins.[4] Although its overall genome similarity is lower to SARS‐CoV‐2 than that of RaTG13, the MP789 pangolin strain isolated from GD pangolins has an almost identical RBD to that of SARS‐CoV‐2. Indeed, pangolin CoVs and SARS‐CoV‐2 possess identical amino acids at the five critical residues of the RBD, whereas RaTG13 only shares one amino acid with SARS‐CoV‐2.[35] ACE2 sequence similarity is higher between humans and pangolins than between humans and bats. Intriguingly, the spike protein of SARS‐CoV‐2 has a higher predicted binding affinity to human ACE2 receptor than to that of pangolins and bats.ix Before the SARS‐CoV‐2 outbreak, pangolins were the only mammals other than bats documented to carry and be infected by SARS‐CoV‐2 related CoV.[12] Recombination events between the RBD of CoV from pangolins and RaTG13‐like backbone could have produced SARS‐CoV‐2 as chimeric strain. For such recombination to occur naturally, the two viruses must have infected the same cell in the same organism simultaneously, a rather improbable event considering the low population density of pangolins and the scarce presence of CoVs in their natural populations.x Moreover, receptor binding studies of reconstituted RaTG13 showed that it does not bind to pangolin ACE2.xi
- Die Möglichkeit, dass Pangoline der Zwischenwirt für SARS-CoV-2 sein könnten, wird seit langem diskutiert. [32-34] Die größte Divergenz zwischen SARS ‐ CoV ‐ 2 und RaTG13 wird in der RBD ihrer Spikeproteine beobachtet. Obwohl die Ähnlichkeit des Gesamtgenoms mit SARS-CoV-2 geringer ist als die von RaTG13, weist der aus GD-Pangolinen isolierte Pangolin-Typ MP789 eine fast identische RBD zu SARS-CoV-2 auf. Tatsächlich besitzen Pangolin-CoVs und SARS-CoV-2 identische Aminosäuren an den fünf kritischen Resten der RBD, während RaTG13 nur eine Aminosäure mit SARS-CoV-2 teilt. [35] Die Ähnlichkeit der ACE2-Sequenzen ist zwischen Menschen und Pangolinen höher als zwischen Menschen und Fledermäusen. Interessanterweise weist das Spike-Protein von SARS-CoV-2 eine höhere vorhergesagte Bindungsaffinität zum menschlichen ACE2-Rezeptor auf als das von Pangolinen und Fledermäusen.ix Vor dem SARS-CoV-2-Ausbruch waren Pangoline die einzigen Säugetiere außer Fledermäusen, deren Nachweis dokumentiert wurde und durch SARS-CoV-2-CoV infiziert sein. [12] Rekombinationsereignisse zwischen der RBD von CoV aus Pangolinen und dem RaTG13-ähnlichen Rückgrat könnten SARS-CoV-2 als chimären Stamm erzeugt haben. Damit eine solche Rekombination auf natürliche Weise stattfinden kann, müssen die beiden Viren gleichzeitig dieselbe Zelle im selben Organismus infiziert haben, was angesichts der geringen Populationsdichte von Pangolinen und des geringen Vorhandenseins von CoVs in ihren natürlichen Populationen eher unwahrscheinlich ist.x Darüber hinaus wurden Rezeptorbindungsstudien durchgeführt von rekonstituiertem RaTG13 zeigte, dass es nicht an Pangolin ACE2.xi bindet
THE FURIN CLEAVAGE SITE: THE KEY DIFFERENCE BETWEEN SARS‐COV‐2 AND ITS CLOSEST RELATIVE RATG13
DIE FURIN CLEAVAGE-STELLE: DER WESENTLICHE UNTERSCHIED ZWISCHEN SARS-COV-2 UND SEINER NÄCHSTEN RELATIVEN RATG13
Doch es gibt nicht nur Gemeinsamkeiten, sondern auch Unterschiede und diese Unterschiede haben gemäß Frau Segreto die Eigenschaft, auf natürlichem Weg praktisch nicht entstehen zu können.
- SARS‐CoV‐2 differs from its closest relative RaTG13 by a few key characteristics. The most striking difference is the acquisition in the spike protein of SARS‐CoV‐2 of a cleavage site activated by a host‐cell enzyme furin, previously not identified in other beta‐CoVs of lineage b[36] and similar to that of Middle East respiratory syndrome (MERS) coronavirus.[35] Host protease processing plays a pivotal role as a species and tissue barrier and engineering of the cleavage sites of CoV spike proteins modifies virus tropism and virulence.[37] The ubiquitous expression of furin in different organs and tissues have conferred to SARS‐CoV‐2 the ability to infect organs usually invulnerable to other CoVs, leading to systemic infection in the body.[38] Cell‐cultured SARS‐CoV‐2 that was missing the above‐mentioned cleavage site caused attenuated symptoms in infected hamsters,[39] and mutagenesis studies have confirmed that the polybasic furin site is essential for SARS‐CoV‐2′s ability to infect human lung cells.[40]
- SARS-CoV-2 unterscheidet sich von seinem nächsten relativen RaTG13 durch einige Schlüsselmerkmale. Der auffälligste Unterschied ist der Erwerb einer durch ein Wirtszellenenzym Furin aktivierten Spaltstelle im Spike-Protein von SARS-CoV-2, die so zuvor noch nie in anderen Beta-CoVs der Linie b identifiziert werden konnte [36] und der vom Middle East Respiratory Syndrome (MERS) des Coronavirus ähnlich ist [35]. Die Verarbeitung von Wirtsproteasen spielt eine zentrale Rolle als Spezies- und Gewebebarriere, und das Engineering der Spaltstellen von CoV-Spike-Proteinen verändert den Virustropismus und die Virulenz. Die allgegenwärtige Expression von Furin in verschiedenen Organen und Geweben hat SARS-CoV-2 die Fähigkeit verliehen, Organe zu infizieren, die normalerweise für andere CoVs unverwundbar sind, was zu einer systemischen Infektion des Körpers führt. [38] Zellkultiviertes SARS-CoV-2, dem die oben genannte Spaltstelle fehlte, verursachte bei infizierten Hamstern abgeschwächte Symptome [39], und Mutagenesestudien haben bestätigt, dass die polybasische Furinstelle für die Infektionsfähigkeit von SARS-CoV-2 essentiell ist, menschliche Lungenzellen zu infizieren. [40]
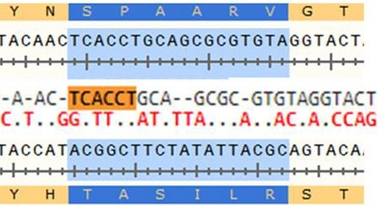
Die Beschreibung der Furin-Stelle
- The polybasic furin site in SARS‐CoV‐2 was created by a 12‐nucleotide insert TCCTCGGCGGGC coding for a PRRA amino acid sequence at the S1/S2 junction (Figure 1). Interestingly, the two joint arginines are coded by two CGGCGG codons, which are rare for these viruses: only 5% of arginines are coded by CGG in SARS‐CoV‐2 or RaTG13, and CGGCGG in the new insert is the only doubled instance of this codon in SARS‐CoV‐2. The CGGCGG insert includes a FauI restriction site, of which there are six instances in SARS‐CoV‐2 and four instances in RaTG13 (and two in MP789). The serendipitous location of the FauI site could allow using restriction fragment length polymorphism (RFLP) techniques [41] for cloning [42] or screening for mutations, [43] as the new furin site is prone to deletions in vitro.[39, 44]
- Die polybasische Furinstelle in SARS-CoV-2 wurde durch ein 12-Nucleotid-Insert TCCTCGGCGGGC erzeugt, das für eine PRRA-Aminosäuresequenz am S1 / S2-Übergang kodiert (Abbildung 1). Interessanterweise werden die beiden gemeinsamen Arginine von zwei CGGCGG-Codons codiert, die für diese Viren selten sind: Nur 5% der Arginine werden von CGG in SARS-CoV-2 oder RaTG13 codiert, und CGGCGG im neuen Insert ist die einzige doppelte Instanz von dieses Codon in SARS-CoV-2. Das CGGCGG-Insert enthält eine FauI-Restriktionsstelle, von der es sechs Instanzen in SARS-CoV-2 und vier Instanzen in RaTG13 gibt (und zwei in MP789). Die zufällige Lage der FauI-Stelle könnte die Verwendung von RFLP-Techniken (Restriktionsfragmentlängenpolymorphismus) [41] zur Klonierung [42] oder zum Screening auf Mutationen [43] ermöglichen, da die neue Furinstelle in vitro zu Deletionen neigt [39, 44 ]]

FIGURE 1
- A study by Zhou et al.[45] reported the discovery of a novel CoV strain RmYN02, which the authors claim exhibits natural PAA amino acid insertions at the S1/S2 cleavage site where SARS‐CoV‐2 has the PRRA insertion. However, upon close examination of the underlying nucleotide sequence of RmYN02 in comparison with its closest ancestors bat‐SL‐CoVZC45 and bat‐SL‐CoVZXC21, no insertions are apparent, just nucleotide mutations (Figure 2).
- Eine Studie von Zhou et al. [45] berichtetet über die Entdeckung eines neuen CoV-Stammes RmYN02, von dem die Autoren behaupten, dass er natürliche PAA-Aminosäure-Insertionen an der S1 / S2-Spaltstelle aufweist, an der SARS-CoV-2 die PRRA-Insertion aufweist. Bei genauer Untersuchung der zugrunde liegenden Nukleotidsequenz von RmYN02 im Vergleich zu seinen engsten Vorfahren bat-SL-CoVZC45 und bat-SL-CoVZXC21 sind jedoch keine Insertionen erkennbar, sondern nur Nukleotidmutationen (Abbildung 2).

FIGURE 2
- Therefore, SARS‐CoV‐2 remains unique among its beta CoV relatives not only due to a polybasic furin site at the S1/S2 junction, but also due to the four amino acid insert PRRA that had created it. The insertion causes a split in the original codon for serine (TCA) in MP789 or RaTG13 to give part of a new codon for serine (TCT) and part of the amino acid alanine (GCA) in SARS‐CoV‐2 (Figure 3).
- Daher bleibt SARS-CoV-2 unter seinen Beta-CoV-Verwandten einzigartig, nicht nur aufgrund einer polybasischen Furinstelle an der S1 / S2-Verbindung, sondern auch aufgrund des PRRA mit vier Aminosäuren, das es erzeugt hat. Die Insertion bewirkt eine Spaltung des ursprünglichen Codons für Serin (TCA) in MP789 oder RaTG13, um einen Teil eines neuen Codons für Serin (TCT) und einen Teil der Aminosäure Alanin (GCA) in SARS-CoV-2 zu erhalten (Abbildung 3). .

FIGURE 3
- The insertion of the furin cleavage site in SARS‐CoV‐2 is not in frame with the rest of the sequence, when compared with the MP789 and the RaTG13 sequences (Figure 3). Therefore, it is possible to exclude that such insertion could have originated by polymerase slippage or by releasing and repriming, because insertion mutations generated by these mechanisms have been postulated to maintain the reading frame of the viral sequence.[46] The possibility that the furin cleavage site could have been acquired by recombination has been recently questioned by Seyran et al.,[47] because the SARS‐CoV‐2 spike protein seems to lack any further recombination event in contrast with the recombination model of other CoVs.
- Die Insertion der Furin-Spaltstelle in SARS-CoV-2 steht im Vergleich zu den Sequenzen MP789 und RaTG13 nicht im Rahmen des Restes der Sequenz (Abbildung 3). Daher kann ausgeschlossen werden, dass eine solche Insertion durch Polymerase-Slippage oder durch Freisetzung und Repriming entstanden sein könnte, da durch diese Mechanismen erzeugte Insertionsmutationen postuliert wurden, um den Leserahmen der Virussequenz aufrechtzuerhalten. [46] Die Möglichkeit, dass die Furin-Spaltstelle durch Rekombination erworben worden sein könnte, wurde kürzlich von Seyran et al. In Frage gestellt [47], da dem SARS-CoV-2-Spike-Protein im Gegensatz zum Rekombinationsmodell anderer kein weiteres Rekombinationsereignis zu fehlen scheint CoVs.
.
CRITIQUE OF “THE PROXIMAL ORIGIN OF SARS‐COV‐2″
KRITIK DES „PROXIMALEN URSPRUNGS VON SARS-COV-2“
In ihrer Arbeit entkräftigt das Forscherteam um Frau Segreto auch die Argumente der Anhänger der Natur-Theorie und erinnert daran, daß 2016 die Baric-Gruppe im Experiment ja unter Verwendung des selben SARS-MA15-Rückgrats, wie ein Jahr zuvor weiderholte und unter Verwendung der RBD von Rs3367 (s.o.), einem nahen Verwandten von RsSHC014, der ebenfalls zuvor von WIV in Yunnan gefunden und nach Lebendkultivierung in „WIV1“ umbenannt wurde, je Ergebnisse lieferte:
- Due to the broad‐spectrum of research conducted over almost 20 years on bat SARS‐CoVs justified by their potential to spill over from animal to human,[48] a possible synthetic origin by laboratory engineering of SARS‐CoV‐2 cannot be excluded. The widely cited article of Andersen et al.[2] stated that SARS‐CoV‐2 has most likely a natural origin. The main argument brought by the authors is that the high‐affinity binding of the SARS‐CoV‐2 spike protein to hACE2 could not have been predicted by models based on the RBD of SARS‐CoV. Based on the structural analysis conducted by Wan et al.,[49] SARS‐CoV‐2 has the potential to recognize hACE2 more efficiently than the SARS‐CoV, which emerged in 2002. Moreover, generation of CoV chimeric strains has recently demonstrated that bat CoV spikes can bind to the hACE2 receptor with more plasticity than previously predicted.[15] All amino acids in the RBD have been extensively analyzed and new models to predict ACE2 affinity are available.[50] In this regard, BatCoV Rs3367 (99.9% identity to WIV1) has been shown to share with SARS‐CoV‐2 four out of six critical residues in the RBD. Considering that WIV1 was shown to directly bind to hACE2, the same assumption could easily have been made about SARS‐CoV‐2 RBD.[51]
- Aufgrund des breiten Forschungsspektrums, das über fast 20 Jahre an SARS-CoVs von Fledermäusen durchgeführt wurde, die durch ihr Potenzial gerechtfertigt sind, vom Tier auf den Menschen überzugehen, [48] kann ein möglicher synthetischer Ursprung von SARS-CoV-2 durch Labortechnik nicht ausgeschlossen werden. Der vielzitierte Artikel von Andersen et al. [2] stellte fest, dass SARS ‐ CoV ‐ 2 höchstwahrscheinlich einen natürlichen Ursprung hat. Das Hauptargument der Autoren ist, dass die hochrangige Bindung des SARS-CoV-2-Spike-Proteins an hACE2 nicht durch Modelle vorausgesagt werden konnte, die auf der RBD von SARS ‐ CoV basieren. Basierend auf der Strukturanalyse von Wan et al. [49] hat SARS-CoV-2 das Potenzial, hACE2 effizienter zu erkennen als das 2002 entstandene SARS-CoV. Darüber hinaus hat die Erzeugung von chimären CoV-Stämmen dies kürzlich gezeigt Fledermaus-CoV-Spikes können mit mehr Plastizität als zuvor vorhergesagt an den hACE2-Rezeptor binden. [15] Alle Aminosäuren in der RBD wurden umfassend analysiert und neue Modelle zur Vorhersage der ACE2-Affinität sind verfügbar. In dieser Hinsicht hat sich gezeigt, dass BatCoV Rs3367 (99,9% Identität zu WIV1) vier von sechs kritischen Resten in der RBD mit SARS-CoV-2 teilt. In Anbetracht der Tatsache, dass gezeigt wurde, dass WIV1 direkt an hACE2 bindet, könnte die gleiche Annahme leicht für SARS-CoV-2-RBD gemacht worden sein.
Frau Segreto und ihr Forscherteam erinnerten die Anhänger der Natur-Theorie auch daran, daß es ja seit 20 Jahren Forschungen an SARS-Chimären gibt und das das WIV eines der zentralen Forschungsszentren dafür ist:
- As described above, creation of chimeric viruses has been carried out over the years with the purpose of studying the potential pathogenicity of bat CoVs for humans. In this context, SARS‐CoV‐2 could have been synthesized by combining a backbone similar to RaTG13 with the RBD of CoV similar to the one recently isolated from pangolins[12], because the latter is characterized by a higher affinity with the hACE2 receptor. Such research could have aimed to identify pangolins as possible intermediate hosts for bat‐CoV potentially pathogenic for humans. Subsequent serial cell or animal passage, as described by Sirotkin & Sirotkin [1] could have provided the perfect adaptation of the RBD to the hACE2.
- Wie oben beschrieben, wurde im Laufe der Jahre die Erzeugung von chimären Viren durchgeführt, um die potenzielle Pathogenität von Fledermaus-CoVs für den Menschen zu untersuchen. In diesem Zusammenhang könnte SARS-CoV-2 durch Kombination eines RaTG13-ähnlichen Rückgrats mit der RBD von CoV ähnlich dem kürzlich aus Pangolinen isolierten synthetisiert worden sein [12], da letzteres durch eine höhere Affinität zum hACE2-Rezeptor gekennzeichnet ist . Diese Forschung hätte darauf abzielen können, Pangoline als mögliche Zwischenwirte für Fledermaus-CoV zu identifizieren, die möglicherweise für den Menschen pathogen sind. Eine nachfolgende serielle Zell- oder Tierpassage, wie von Sirotkin & Sirotkin [1] beschrieben, hätte die perfekte Anpassung der RBD an die hACE2 ermöglichen können.
Das Forscherteam um Frau Segreto erinnerte darüber hinaus auch daran, daß die Arbeiten von Andersen et al. durch neue Erkenntnisse überholt sind:
- Regarding the furin cleavage site, Andersen et al.[2] state that “the functional consequence of the polybasic cleavage site in SARS‐CoV‐2 is unknown.” New studies from several groups have lately identified this activation site as possibly enabling the virus to spread efficiently between humans and attack multiple organs.[52] Experiments on proteolytic cleavage of CoV spike proteins have been recently suggested as future key studies to understand virus transmissibility in different hosts.[50]
- In Bezug auf die Furinspaltungsstelle haben Andersen et al. [2] festgestellt, dass „die funktionelle Konsequenz der polybasischen Spaltstelle in SARS-CoV-2 unbekannt ist“. Neue Studien aus mehreren Gruppen haben kürzlich festgestellt, dass diese Aktivierungsstelle möglicherweise eine effiziente Ausbreitung des Virus zwischen Menschen und den Angriff auf mehrere Organe ermöglicht. [52] Experimente zur proteolytischen Spaltung von CoV-Spike-Proteinen wurden kürzlich als zukünftige Schlüsselstudien vorgeschlagen, um die Virusübertragbarkeit in verschiedenen Wirten zu verstehen. [50]
Dies betrifft insbesondere den Umfang des durch die jahrzehntelange Forschung zur Verfügung stehenden Materials, den Andersen unterschätzen dürfte:
- Andersen et al.[2] also state, based on the work of Almazan et al.[53] that “the genetic data irrefutably show that SARS‐CoV‐2 is not derived from any previously used virus backbone.” In the last 6 years before the outbreak of SARS‐CoV‐2 the number of potential bat backbones has been undeniably increased by several bat CoV screenings, last but not least bringing RaTG13 to scientific attention in January 2020. Other possible backbones could, as well, still wait for publication.
- Andersen et al. [2] auch angeben, basierend auf der Arbeit von Almazan et al. [53] dass „die genetischen Daten unwiderlegbar zeigen, dass SARS-CoV-2 nicht von einem zuvor verwendeten Virus-Backbone abgeleitet ist.“ In den letzten 6 Jahren vor dem Ausbruch von SARS-CoV-2 wurde die Anzahl potenzieller Fledermaus-Rückgrate unbestreitbar durch mehrere Fledermaus-CoV-Screenings erhöht, was nicht zuletzt RaTG13 im Januar 2020 wissenschaftlich bekannt machte. Andere mögliche Rückgrate könnten könnten noch immer auf ihre Veröffentlichung warten.
Und sie wirft Andersen vor denkbare Entstehungswege einfach ideologisch von vorne herein ausgeschlossen zu haben:
- Andersen et al.[2] affirm that “the acquisition of both the polybasic cleavage site and predicted O‐linked glycans also argues against culture‐based scenarios.” Methods for insertion of a polybasic cleavage site in infectious bronchitis CoV are given in Cheng et al.[54] and resulted in increased pathogenicity. Concerning the predicted O‐linked glycans around the newly inserted polybasic site, it should be noted that this prediction was not confirmed by Cryo‐EM inquiry into the SARS‐CoV‐2 spike glycoprotein.[55] Nevertheless, while it is true that O‐linked glycans are much more likely to arise under immune selection, they could be added in the lab through site‐directed mutagenesis[56] or arise in the course of in vivo experiments, for example, in BLT‐L mice with human lung implants and autologous human immune system[57] or in mice expressing the hACE2 receptor.[31] To overcome problems of bat CoV isolation, experiments based on direct inoculation of bat CoV in suckling rats have been carried out.[58] Humanized mice, ferrets, primates and/or other animals with similar ACE2 conformation could have all been used for serial passage experiments, as described in detail by Sirotkin and Sirotkin.[1]
- Andersen et al. [2] geben zu, dass „die Akquisition sowohl der polybasischen Spaltungsstelle als auch vorhergesagte O-verknüpfte Glykans auch gegen kulturbasierte Szenarien argumentieren.“ Methoden zur Insertion einer polybasischen Spaltstelle bei infektiöser Bronchitis CoV werden wiederum durch Cheng et al.[54] gezeigt und führten zu einer erhöhten Pathogenität. In Bezug auf die vorhergesagten O-verknüpften Glykane um die neu eingefügte polybasische Stelle sollte beachtet werden, dass diese Prognose nicht durch eine Cryo-EM-Untersuchung des SARS-CoV-2-Spike-Glykoproteins bestätigt wurde. Obwohl es wahr ist, dass O-verknüpfte Glykane unter Immunselektion viel wahrscheinlicher auftreten, könnten sie aber auch im Labor durch ortsgerichtete Mutagenese hinzugefügt werden [56] oder im Verlauf von In-vivo-Experimenten entstehen, so, we es z.B. bei BLT-L-Mäusen mit menschlichen Lungenimplantaten und autologem menschlichem Immunsystem [57] der FAll ist, oder in Mäusen mit dem hACE2-Rezeptor [31]. Um wiederum die Probleme der Fledermaus-CoV-Isolierung zu überwinden, wurden Experimente durchgeführt, die auf der direkten Inokulation von Fledermaus-CoV bei säugenden Ratten beruhen. Humanisierte Mäuse, Frettchen, Primaten und / oder andere Tiere mit ähnlicher ACE2-Konformation könnten alle für serielle Passageexperimente verwendet worden sein, wie es auch von Sirotkin und Sirotkin ausführlich beschrieben wurde.
Auch ist der Schluß Andersens, daß Forschungsschritte nicht beschrieben wurden damit gleichzusetzen sei, daß Forschungen nicht getätigt wurden.
- Andersen et al.[2] also state that “subsequent generation of a polybasic cleavage site would have then required repeated passage in cell culture or animals with ACE2 receptors similar to those of humans, but such work has also not previously been described.” It should not be excluded that such experiments could have been aborted due to the SARS‐CoV‐2 outbreak, before a possible publication of the results or that the results were never intended to be published.
- Andersen et al. [2] stellen auch fest, dass „die nachfolgende Erzeugung einer polybasischen Spaltstelle dann eine wiederholte Passage in Zellkultur oder Tieren mit ACE2-Rezeptoren ähnlich denen von Menschen erfordert hätte, aber solche Arbeiten wurden auch bisher nicht beschrieben.“ Es sollte nicht ausgeschlossen werden, dass solche Experimente aufgrund des SARS-CoV-2-Ausbruchs vor einer möglichen Veröffentlichung der Ergebnisse abgebrochen wurden oder dass die Ergebnisse niemals veröffentlicht werden sollten.
Andersen übersieht in seinen Arbeiten außerdem, daß RaTG13 gezeigt hat, daß RaTG13 gar nicht in der Lage ist, an SARS-Viren aus Pangolinen zubinden und daher logisch notwendig gar nicht in der Natur entstanden sein kann:
- It is important to mention that RaTG13 and the pangolin CoV sequences from smuggled pangolins confiscated in the GD province in March 2019, and to which most of published papers supporting a natural origin of SARS‐CoV‐2 refer,[2] have recently been questioned as to the accuracy of their assembly dataxii and require further analyses to prove their correctness.[xiii ,xiv ] It should also be noted that in vitro receptor binding studies of reconstituted RaTG13 yielded some peculiar results.[xi] The most surprising observation was that RaTG13, unlike SARS‐CoV‐2, is unable to bind ACE2 in R. macrotis bats, a close relative of RaTG13’s purported host, R. affinis[59] (whose ACE2 receptor has not yet been tested). At the same time, RaTG13 was observed to bind hACE2[60], but not as well as ACE2 of rats and mice, to which SARS‐CoV‐2 did not bind at all. Is it possible that just as SARS‐MA15 was a mouse‐adapted strain of SARS, RaTG13 is actually a mouse‐adapted version of a CoV extracted from the Mojiang cave, rather than a strain obtained from a bat fecal swab? Unfortunately, the RaTG13 sample has been exhausted and it is no longer available for external examination,[11] which is unfortunate given a number of inconsistencies in its sequencing raw data. Also, the status and availability of the Mojiang miners’ samples remain as well an open and highly relevant question. Several samples from the miners have been collected[7, 8] and likely stored, and it would be of great value to test them for the presence of SARS‐CoV‐2‐like CoVs.
- Es ist an dieser Stelle auch wichtig darauf aufmerksam zu machen, dass RaTG13 und die Pangolin-CoV-Sequenzen von geschmuggelten Pangolinen stammen, die im März 2019 in der GD-Provinz beschlagnahmt wurden und auf die sich die meisten veröffentlichten Artikel beziehen, die einen natürlichen Ursprung von SARS-CoV-2 belegen wollen, [2] aber kürzlich in Frage gestellt wurden hinsichtlich der Genauigkeit ihrer Assemblierungsdaten, was weitere Analysen erfordert, um deren Richtigkeit zu beweisen. [xiii, xiv] Es sollte auch beachtet werden, dass In-vitro-Rezeptorbindungsstudien von rekonstituiertem RaTG13 einige besondere Ergebnisse erbrachten. [xi] Die überraschendste Beobachtung war die, daß RaTG13 ist im Gegensatz zu SARS-CoV-2 nicht in der Lage war, ACE2 in R. macrotis-Fledermäusen zu binden, einem nahen Verwandten von RaTG13s angeblichem Wirt R. affinis [59] (dessen ACE2-Rezeptor noch nicht getestet wurde). Gleichzeitig wurde beobachtet, dass RaTG13 hACE2 bindet, aber nicht so gut wie ACE2 von Ratten und Mäusen, an die SARS-CoV-2 überhaupt nicht bindet. Ist es also möglich, dass RaTG13 genau wie SARS-MA15 ein mausangepasster SARS-Stamm ist, tatsächlich eine mausangepasste Version eines aus der Mojiang-Höhle extrahierten CoV ist, und kein Stamm, der aus einem Fledermaus-Kotabstrich gewonnen wurde? Leider ist die RaTG13-Probe erschöpft und steht nicht mehr für externe Untersuchungen zur Verfügung [11], was angesichts einer Reihe von Inkonsistenzen bei der Sequenzierung der Rohdaten bedauerlich ist. Auch der Status und die Verfügbarkeit der Proben der Bergleute in Mojiang bleiben eine offene und hochrelevante Frage. Mehrere Proben der Bergleute wurden gesammelt [7, 8] und wahrscheinlich gelagert, und es wäre von großem Wert, sie auf das Vorhandensein von SARS-CoV-2-ähnlichen CoVs zu testen.
Frau Segreto macht außerdem auf merkwürdige Vorkommnisse um Frau Shi und ihre Arbeiten aufmerksam, wie z.B. verschwundene Datenbanken:
- Another open question is the reason for modification and subsequent deletion of WIV’s own viral database. In May 2020, several media outlets have reported that the change tracking system of WIV’s internal database showed that the database was renamed from “Wildlife‐borne viral pathogen database” to “Bat and rodent‐borne viral pathogen database,” and its description was edited to replace instances of “wild animal” by “bat and rodent”; in addition, mention of “arthropod vectors” was deleted.xv The database description reported that it contained over 60 Mb of data in structured query language (SQL) format, but at as of early May 2020 the download link no longer worked.xvi Subsequently, the database page was taken down in its entirety but its snapshot is still available on Web Archive.xvii It is possible that other international CoV labs might have downloaded the SQL archive of the WIV database before it was taken down, in which case such groups should make those data publicly available.
- Eine weitere offene Frage ist der Grund für die Änderung und anschließende Löschung der eigenen Virendatenbank von WIV. Im Mai 2020 haben mehrere Medien berichtet, dass das Änderungsverfolgungssystem der internen Datenbank von WIV gezeigt hat, dass die Datenbank von „Datenbank für durch Viren übertragene Viren“ in „Datenbank mit durch Viren übertragenen Fledermäusen und Nagetieren“ umbenannt und ihre Beschreibung dahingehend bearbeitet wurde, Fälle von „wildem Tier“ durch „Fledermaus und Nagetier“ zu ersetzen; Darüber hinaus wurde die Erwähnung von „Arthropodenvektoren“ gelöscht.xv In der Datenbankbeschreibung wurde angegeben, dass sie mehr als 60 MB Daten im SQL-Format (Structured Query Language) enthielt. Ab Anfang Mai 2020 funktionierte der Download-Link jedoch nicht mehr.xvi Anschließend Die Datenbankseite wurde vollständig entfernt, der Snapshot ist jedoch weiterhin im Web Archive.xvii verfügbar. Möglicherweise haben andere internationale CoV-Labors das SQL-Archiv der WIV-Datenbank heruntergeladen, bevor sie entfernt wurde. In diesem Fall sind solche Gruppen sollten diese Daten öffentlich zugänglich machen.
Im Mai 2020 verschwinden in China Menschen und Daten und ganze Datenbanken
.
HOW COULD THE VIRUS HAVE ESCAPED FROM A LAB?
Historische Fälle, wie Viren aus Laboren entwichen
All diese Argumente müssen außerdem vor dem Hintergrund der Tatsache bewertet werden, daß China seit Jahrzehnten an Fledermaus-SARS-Viren forscht:
- The leak of highly dangerous pathogens from laboratories is not a rare event and occurrences have been documented in several countries. The most notable lab leak known is the 1977 H1N1 lab escape from China that caused a worldwide pandemic.[61] The most recent one is the November 2019 outbreak of brucellosis that occurred in two research centers in Lanzhou, China, infecting over 100 students and staff members.[62] Several lab escapes of the first SARS virus have been reported as well: in the summer of 2003 in Singapore,[63] then in December 2003 in Taiwan,xviii and in the spring of 2004 twice in China.xix
- Das Austreten hochgefährlicher Krankheitserreger aus Laboratorien ist kein seltenes Ereignis, und in mehreren Ländern wurden Vorkommen dokumentiert. Das bemerkenswerteste bekannte Laborleck ist die Flucht des H1N1-Labors aus China im Jahr 1977, die eine weltweite Pandemie auslöste. [61] Der jüngste ist der Ausbruch der Brucellose im November 2019, der in zwei Forschungszentren in Lanzhou, China, auftrat und über 100 Studenten und Mitarbeiter infizierte. [62] Es wurden auch mehrere Laborfluchten des ersten SARS-Virus gemeldet: im Sommer 2003 in Singapur [63], dann im Dezember 2003 in Taiwan, xviii und im Frühjahr 2004 zweimal in China.xix
Von Anbeginn an gab es Bedenken über die Sicherheit im WIV:
- Concerns about WIV’s lab safety were raised in 2018 by U.S. Embassy officials after visiting the Institute and having an interview with Zhengli Shi. The lab auditors summarized their worries in subsequent diplomatic cables to Washington.xx Chinese experts have also raised concerns about lab safety in their own country, lamenting that “lab trash can contain man‐made viruses, bacteria or microbes” and that “some researchers discharge laboratory materials into the sewer after experiments without a specific biological disposal mechanism.”xxi
- Die Beamten der US-Botschaft äußerten 2018 Bedenken hinsichtlich der Laborsicherheit von WIV, nachdem sie das Institut besucht und ein Interview mit Zhengli Shi geführt hatten. Die Laborprüfer fassten ihre Sorgen in nachfolgenden diplomatischen Noten nach Washington.xx zusammen. Chinesische Experten äußerten ebenfalls Bedenken hinsichtlich der Laborsicherheit in ihrem eigenen Land und beklagten, dass „Laborabfälle künstliche Viren, Bakterien oder Mikroben enthalten können“ und dass „einige Forscher Labormaterial nach Experimenten in den Abwasserkanal entworgen, und zwar ohne spezifische biologische Entsorgungsregeln zu beachten. “xxi
Schwierigkeiten mit der Sicherheit sind sogar ein Dauerproblem:
- American labs have also had their share of safety issues. Recently, research operations in the Biosafety level (BSL)‐4 United States Army Medical Research Institute of Infectious Diseases (USAMRIID) facility in Fort Detrick were interrupted in August 2019 following safety violations, in particular, relating to the disposal of infective materials.xxii Other US labs have been cited for safety issues as well.22]
- Amerikanische Labore hatten auch ihren Anteil an Sicherheitsproblemen. Vor kurzem wurden im August 2019 Forschungsarbeiten in der Einrichtung des US-amerikanischen medizinischen Forschungsinstituts für Infektionskrankheiten (USAMRIID) der Biosicherheitsstufe (BSL) -4 in Fort Detrick unterbrochen, insbesondere im Zusammenhang mit der Entsorgung von infektiösem Material.xxii Andere US-Labors wurden ebenfalls aus Sicherheitsgründen angeführt.22]
Von daher bleiben eine Vielzahl an Möglichkeiten, wie ein im WIV künstlich hergestelltes Covid-19-Virus theoretisch hätte nach draußen entkommen können:
- A number of scenarios causing SARS‐CoV‐2 to leak from a lab can be hypothesized. For example, an infected animal could have escaped from a lab or it could have scratched or bitten a worker (a concern raised in 2017 about the establishment of a BSL‐4 primate vaccine testing facility in Kunming, Yunnan[64]), or a researcher could have accidentally stuck themselves with inoculate (as happened in two cases in Russiaxxiii ). Until 2020, CoVs were not considered particularly deadly or virulent. SARS‐like CoVs did not require BSL‐4 and could be manipulated under BSL‐2 and BSL‐3[42] conditions, making an accidental leak more likely. Aerosol experiments with CoVs[65] could result in lab leak as well, because a failure in the equipment used could go unnoticed for a long time before infection of lab workers is detected. Finally, the virus could potentially have leaked through the sewage system if proper waste disposal and/or decontamination procedures were not followed.
- Es kann eine Reihe von Szenarien angenommen werden, bei denen SARS-CoV-2 aus einem Labor austritt. Zum Beispiel könnte ein infiziertes Tier aus einem Labor entkommen sein oder einen Arbeiter zerkratzt oder gebissen haben (eine 2017 geäußerte Besorgnis über die Einrichtung einer BSL-4-Testanlage für Primatenimpfstoffe in Kunming, Yunnan [64]) oder a Forscher hätten sich versehentlich mit Impfungen festhalten können (wie in zwei Fällen in Russlandxxiii). Bis 2020 galten CoVs nicht als besonders tödlich oder virulent. SARS-ähnliche CoVs erforderten kein BSL-4 und konnten unter BSL-2- und BSL-3-Bedingungen [42] manipuliert werden, wodurch ein versehentliches Leck wahrscheinlicher wurde. Aerosolexperimente mit CoVs [65] können ebenfalls zu Laborlecks führen, da ein Ausfall der verwendeten Geräte lange Zeit unbemerkt bleiben kann, bevor eine Infektion der Laboranten festgestellt wird. Schließlich könnte das Virus möglicherweise durch das Abwassersystem gelangt sein, wenn die ordnungsgemäße Entsorgung und / oder Dekontamination nicht befolgt worden wäre.
.
CONCLUSIONS AND OUTLOOK
SCHLUSSFOLGERUNGEN UND AUSBLICK
Die künstliche Herstellung von Covid-19 ist wissenschaftlich möglich:
- On the basis of our analysis, an artificial origin of SARS‐CoV‐2 is not a baseless conspiracy theory that is to be condemned[66] and researchers have the responsibility to consider all possible causes for SARS‐CoV‐2 emergence. The insertion of human‐adapted pangolin CoV RBD obtained by cell/animal serial passage and furin cleavage site could arise from site‐directed mutagenesis experiments, in a context of evolutionary studies or development of pan‐CoV vaccines or drugs. A recent article in Nature[67] affirms that a laboratory origin for SARS‐CoV‐2 cannot be ruled out, as researchers could have been infected accidentally, and that gain‐of‐function experiments resulting in SARS‐CoV‐2 could have been performed at WIV. Genetic manipulation of SARS‐CoV‐2 may have been carried out in any laboratory in the world with access to the backbone sequence and the necessary equipment and it would not leave any trace. Modern technologies based on synthetic genetics platforms allow the reconstruction of viruses based on their genomic sequence, without the need of a natural isolate.[68]
- Auf der Grundlage unserer Analyse ist ein künstlicher Ursprung von SARS-CoV-2 keine substanzlose Verschwörungstheorie, die verurteilt werden muss [66], und die Forscher haben die Verantwortung, alle möglichen Ursachen für die Entstehung von SARS-CoV-2 zu berücksichtigen. Die Insertion von an den Menschen angepasstes Pangolin-CoV-RBD, das durch serielle Passage von Zellen / Tieren und Furin-Spaltungsstelle erhalten wurde, könnte auch aus ortsgebundenen Mutagenese-Experimenten im Zusammenhang mit evolutionären Studien oder Entwicklung von Pan-CoV-Impfstoffen oder Medikamenten entstehen. Ein kürzlich in Nature [67] veröffentlichter Artikel bestätigt, dass ein Laborursprung für SARS-CoV-2 nicht ausgeschlossen werden kann, weil Forscher versehentlich infiziert worden sein könnten und dass Experimente mit Funktionsgewinn, am WIV könnten durchgeführt worden sein, die dann letztendlich zu SARS-CoV-2 führten. Die genetische Manipulation von SARS ‐ CoV ‐ 2 wurde möglicherweise in jedem Labor der Welt mit Zugang zur Backbone-Sequenz und den erforderlichen Geräten durchgeführt und hinterließ daher keine Spuren. Moderne Technologien, die auf synthetischen Genetikplattformen basieren, ermöglichen aber die Rekonstruktion von Viren basierend auf ihrer Genomsequenz, ohne dass ein natürliches Isolat erforderlich ist.
Eine systematische Untersuchung dieser Möglichkeiten ist daher wissenschaftlich auch geboten:
- A thorough investigation on strain collections and research records in all laboratories involved in CoV research before SARS‐CoV‐2 outbreak is urgently needed. Special attention should be paid to strains of CoVs that were generated in virology laboratories but have not yet been published, as those possibly described in the deleted WIV database. Because finding a possible natural host could take years, as with the first SARS,[67] or never succeed, equal priority should be given to investigating natural and laboratory origins of SARS‐CoV‐2. Xiao Qiang, a research scientist at Berkeley, recently stated: “To understand exactly how this virus has originated is critical knowledge for preventing this from happening in the future.”[xxi]
- Eine gründliche Untersuchung der Stammsammlungen und Forschungsaufzeichnungen in allen an der CoV-Forschung beteiligten Labors vor dem Ausbruch von SARS-CoV-2 ist dringend erforderlich. Besondere Aufmerksamkeit sollte CoV-Stämmen gewidmet werden, die in Virologielabors erzeugt, aber noch nicht veröffentlicht wurden, wie sie möglicherweise in der gelöschten WIV-Datenbank beschrieben sind. Da die Suche nach einem möglichen natürlichen Wirt wie beim ersten SARS Jahre dauern kann [67] oder niemals erfolgreich sein kann, sollte der Untersuchung der natürlichen und labortechnischen Herkunft von SARS-CoV-2 die selbe Priorität eingeräumt werden. Xiao Qiang, ein Wissenschaftler in Berkeley, erklärte kürzlich: „Um genau zu verstehen, wie dieses Virus entstanden ist, ist der Schlüssel dazu, so etwas in Zukunft zu verhindern.“ [Xxi]


